SMILES Convolution Fingerprint(SCFP) review
Convolutional neural network based on SMILES representation of compounds for detecting chemical motif, 2018
들어가며
이번에 읽은 논문은 Convolutional neural network based on SMILES representation of compounds for detecting chemical motif review 이다. 해당 논문에서는 CNN을 활용해, 기존의 fingerprint보다 성능이 좋은 새로운 fingerprint 를 정의한다. 논문에 따르면, ECFP( known as morgan fingerprint) 보다도 성능이 좋다고 명시하고 있다. 모델의 학습과 평가에 사용된 데이터는 TOX21 dataset 이며, metric으로는 ROC-AUC score를 활용하였다.
CNN에는 DNA 등 bioinformatic 분야에서 널리 활용되고 있다. 예를 들어, classification of DNA sequence, extraction of a sequence motif, predicting DNA-protein binding 등에 사용되기도 한다. 한 가지 방법으로는, 4가지 DNA nucleotides 들을 one-hot coding representation으로 표현하고 그 데이터를 input 으로 준다. 그 다음 one-dimensional convolution filter which passed to a sequence 가 일종의 motif로 사용되는 것이다. 해당 논문에서 사용한 방법은 이런 방법론을 chemical motif를 찾는 데에 활용한 것이다.
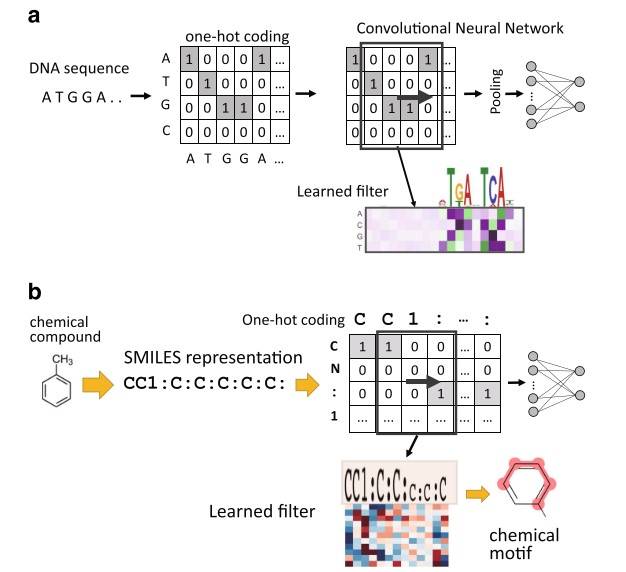
Main Idea
논문에서 강조하고 있는 점은, CNN은 표현 학습으로서 새로운 fingerprint를 찾을 수 있으며, CNN의 kernel로부처 chemical motif 또한 발견해 낼 수 있다는 점이다. 논문의 세부적인 구조는 아래 그림과 같다.
1. Model Architecture
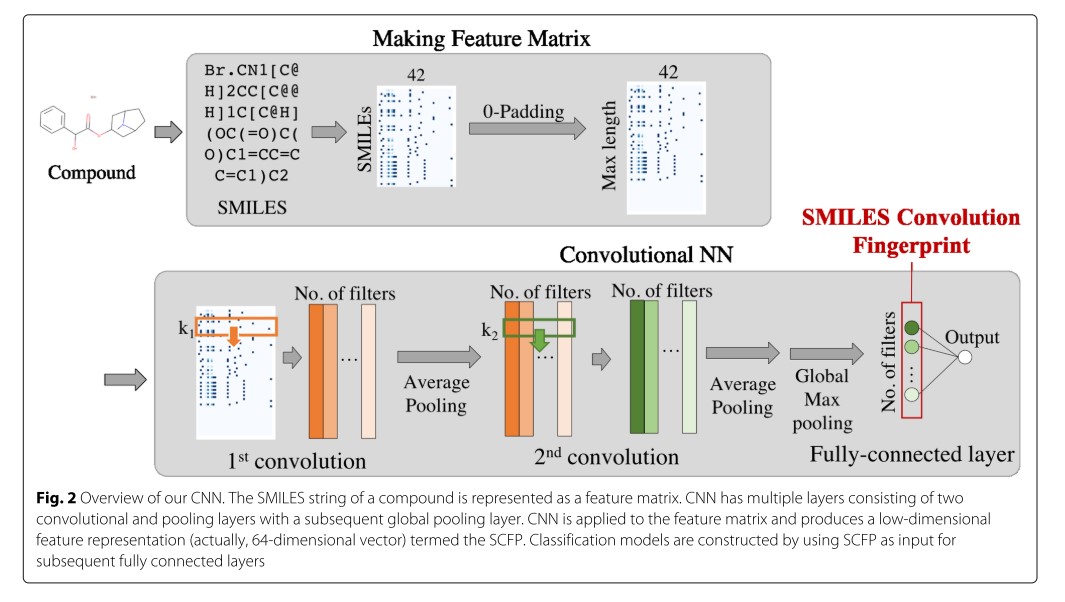
해당 논문에서는 , 기존의 SMILES를 사용하지 않고, one-hot coding을 활용한 SMILES matrix 를 새롭게 만들고 그것을 input data로 활용한다. 그 다음 위의 그림과 같이 2번의 convolution을 거치고 global max pooling 이 후, 결과적으로 64차원의 SMILES Convolution Fingerprint 가 생성된다. 분류기 모델은 data가 입력되어, concolution 구조를 지나 SCFP (SMILES Convolution Fingerprint)가 생성되면 그것을 fully-connected 로 연결하여 output 값을 낸다.
2. SMILES feature matrix
SMILES feature matrix를 구성하고 있는 요소들을 살펴보면 아래 그림과 같다. Table1 과 같다.
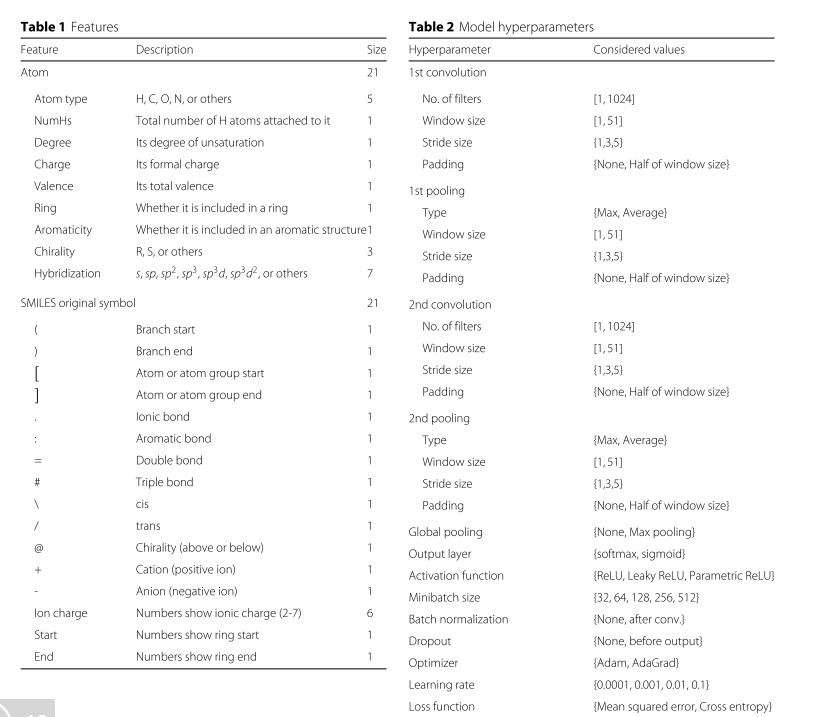
총 42가지의 feature들로 구성되어 있는데, 21가지는 원자에 관한 특성을 나타내는 요소들로 구성되어 있다. 여기에는 원자 type, degree, charge, chirality 등의 요소가 포함된다. 대부분의 요소가 binary 데이터로 표현되지만 feature의 특성상, numerical로 표현할 수 밖에 없는 부분들도 있었다. (degree, chargeand chirality와 같은 부분들) 그런 부분들은 RDkit tool 을 활용해 계산되었다. 나머지 21가지는 기존의 SMILES 형식의 데이터에서 나타나는 symbol들을 구성하고 있다. feature matrix의 길이는 dataset에서 가장 긴 data를 기준으로 정하였으며 나머지 빈 공간은 모두 0으로 padding 하였다. 논문에서 사용된 size는 (400,42) 이다.
CNN의 자세한 hyper parameter는 위의 그림에서 Table2를 보면 자세하게 명시되어 있다.
3. Chemical motif
이번 논문의 장점으로 내세우고 있는 점 중 하나가 바로 chemical motif 부분이다. SCFP가 global max pooling 과정을 거쳐 나오기 때문에, SCFP의 하나의 차원은, 2nd convolution layer의 filter들 중 하나일 것이며 이를 추적해 보면 중요한 substructure 정보를 얻을 수 있을 것이란 것이다.
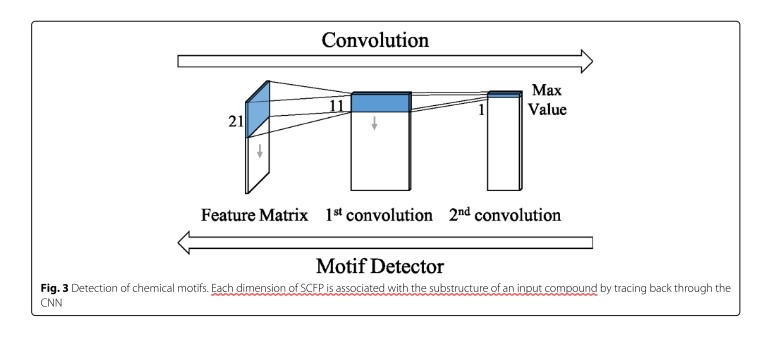
위의 그림은 논문에서 설명하고 있는 network를 tracing back 하여 substructure 와 연관짓는 부분이다. 만약 어떤 SCFP에서 어떤 차원의 값이 크게 기여한다면 그 부분에 대응하는 filter가 있을 것이고, filter가 큰 값을 갖게 한 부분이 substructure로서 중요한 역할을 할 수 있다고 말하고 있다.
한편, SCFP의 모든 차원이 기여하는 바의 scale이 다르기 때문에 어떤 filter가 상대적으로 크게 기여하는지를 파악하기 어렵다. 따라서 normalization을 수행해준다. 1) 모든 SCFP를 계산하고, 2) global max pooling 값을 확인한다. 다음, 3) 모든 compounds의 모든 filter에 대해 mean, variance 를 계산한 뒤, 4) 필터에 대응하는 평균과 분산을 이용하여 SCFP를 각 차원의 Z-score로 변환한다.
결과
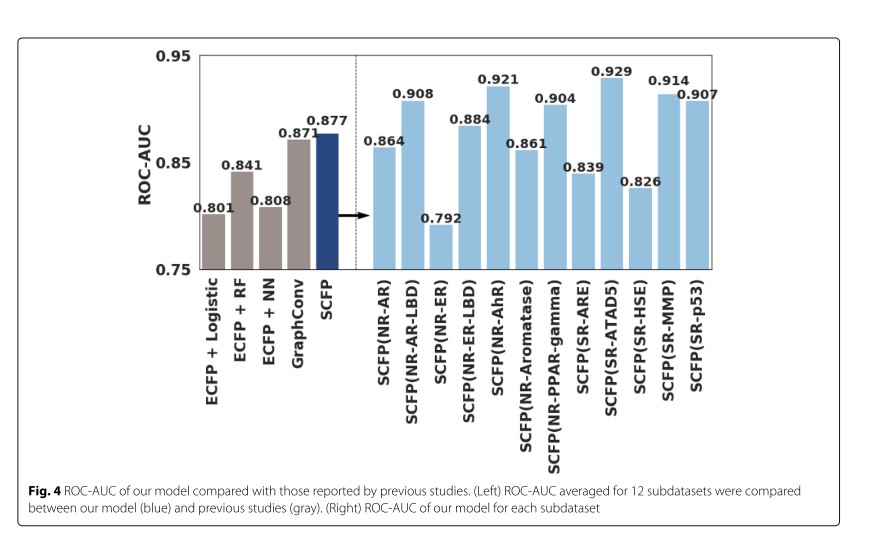 기존의 ECFP를 통해 fingerprint를 추출하여 사용한 것에 비해 SCFP 모델 활용한 모델이 ROC-AUC 에서 더 높은 성능을 보여주고 있는 것을 보여주고 있다. Figure 4의 화살표를 기준으로 오른쪽은 왼쪽의 값에 대한 sub dataset들에 대해 각각의 성능을 표현하고 있다.
기존의 ECFP를 통해 fingerprint를 추출하여 사용한 것에 비해 SCFP 모델 활용한 모델이 ROC-AUC 에서 더 높은 성능을 보여주고 있는 것을 보여주고 있다. Figure 4의 화살표를 기준으로 오른쪽은 왼쪽의 값에 대한 sub dataset들에 대해 각각의 성능을 표현하고 있다.
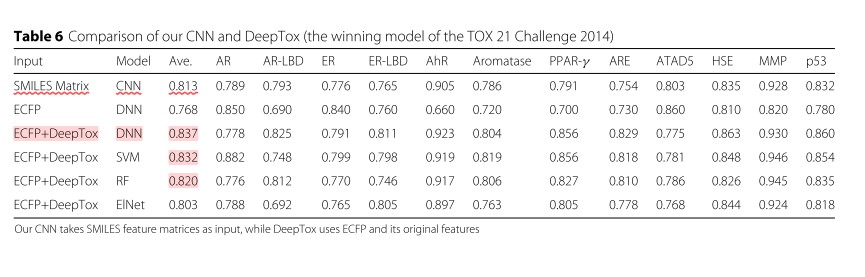 Table 6의 표는 2014년 TOX21에서 우승한 모델인 DeepTox 와 ECFP를 함께 input feature 로 활용한 모델과 논문의 모델에서 얻은 SMILES Matrix 와 CNN을 통한 성능을 비교한 테이블을 보여주고 있다.
Table 6의 표는 2014년 TOX21에서 우승한 모델인 DeepTox 와 ECFP를 함께 input feature 로 활용한 모델과 논문의 모델에서 얻은 SMILES Matrix 와 CNN을 통한 성능을 비교한 테이블을 보여주고 있다.
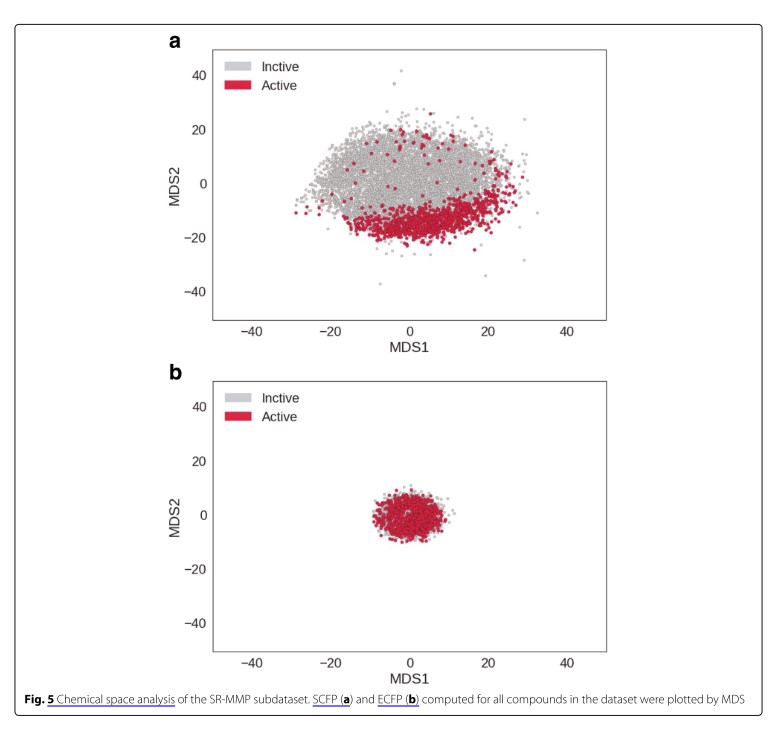 위의 figure 5는 MDS를 통해 그려진 active/inactive에 대한 chemical space를 분석한 Figure이다. a에서 보여지는 결과는 SCFP를 사용한 결과로, active와 inactive가 좀 더 분명하게 나뉘어 있는 것을 볼 수 있다. 반면, b의 ECFP를 통한 결과는 active와 inactive가 겹쳐진 모습을 보여주고 있다.
위의 figure 5는 MDS를 통해 그려진 active/inactive에 대한 chemical space를 분석한 Figure이다. a에서 보여지는 결과는 SCFP를 사용한 결과로, active와 inactive가 좀 더 분명하게 나뉘어 있는 것을 볼 수 있다. 반면, b의 ECFP를 통한 결과는 active와 inactive가 겹쳐진 모습을 보여주고 있다.
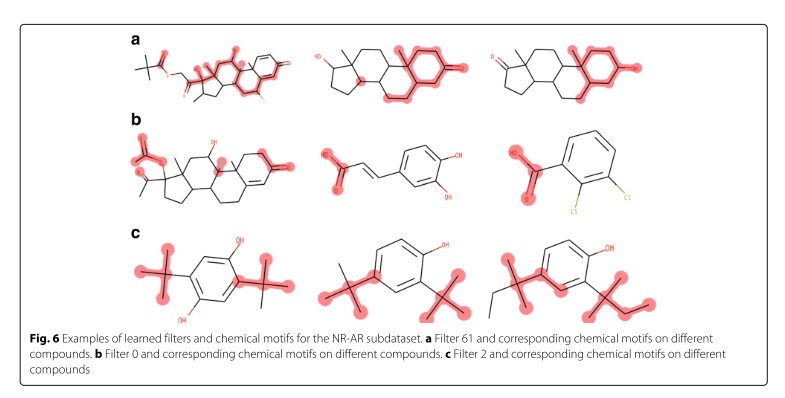 위의 Figure 6는 NR-AR sub dataset에서 특정 filter가 각각의 compound에 대해 chemical motif를 잘 발견하고 있다는 것을 말하며 보여주는 그림이다. a의 그림을 보면 androgen receptor에 binding 되기 위해 중요하게 작용하는 steroid-like chemical motif를 잘 발견하고 있다고 말한다. 또한 다른 detected motif도 androgen receptor를 위한 중요 작용기의 후보군으로 볼 수 있다고 한다. 따라서 이는 classification 뿐만 아니라, drug discovery의 단서를 제공하는 수단으로 작용할 수 있다고 한다.
위의 Figure 6는 NR-AR sub dataset에서 특정 filter가 각각의 compound에 대해 chemical motif를 잘 발견하고 있다는 것을 말하며 보여주는 그림이다. a의 그림을 보면 androgen receptor에 binding 되기 위해 중요하게 작용하는 steroid-like chemical motif를 잘 발견하고 있다고 말한다. 또한 다른 detected motif도 androgen receptor를 위한 중요 작용기의 후보군으로 볼 수 있다고 한다. 따라서 이는 classification 뿐만 아니라, drug discovery의 단서를 제공하는 수단으로 작용할 수 있다고 한다.
마치며
위의 논문이 보여주고 있는 것은 기존의 hand crafted features에 비해 모델이 직접 발견하고 있는 SMILES matrix가 유의미한 성능을 내고 있으며, 또한 해당 feature들로부터 drug discovery의 단서들을 발견할 수 있을 가능성을 제시하고 있는 논문으로 흥미롭게 읽혔다. 딥러닝이 많은 분야에 적용되며 훌륭한 성과를 거두고 있는데, Cheminformatics 분야에서 또한 많은 기대를 가지게 만든다.